Populations de mildiou de la pomme de terre et de la tomate en France en 2024:
caractéristiques et conséquences
L’agent du mildiou de la pomme de terre et de la tomate, Phytophthora infestans, reste une menace majeure pour les agriculteurs et maraichers comme pour les jardiniers amateurs. Présent dans et aux abords des cultures (parcelles de production, jardins de particuliers, tas de déchets avoisinant les champs, repousses), ce parasite est capable d’initier des épidémies à partir de nombreux réservoirs d’inoculum, surtout lorsque la prophylaxie (bâchage ou destruction des tas de déchets et des repousses) est insuffisamment mise en oeuvre. De plus, de nouveaux génotypes ou lignées clonales apparaissent régulièrement qui, devenant parfois invasifs, impactent directement la lutte.
Depuis 2013, un suivi des populations de P. infestans est organisé chaque année en France pour surveiller ces évolutions, grâce à un réseau mobilisant un grand nombre d’acteurs régionaux (réseau BSV, chambres d’agriculture, instituts techniques, producteurs de plants, coopératives, négociants, industriels, CETA, etc…) et au soutien scientifique d’INRAE. Cette épidémiosurveillance repose sur :
- une collecte facilitée d’échantillons biologiques, par simple écrasement d’un tissu symptômatique sur une carte FTA® permettant de fixer et de conserver l’ADN de l’échantillon ;
- une caractérisation génotypique du parasite, à partir de l’ADN contenu sur ces cartes. Ceci fournit l‘empreinte génétique de chaque individu, et donc l’identification des principales lignées clonales et variants nouveaux présents sur le territoire.
Cette méthode d’identification étant partagée au niveau européen, les données françaises peuvent être mises en regard de celles des pays voisins, ce qui permet de mieux tracer et comprendre les changements affectant les populations du parasite et ses voies de dissémination. La compilation à l’échelle européenne est effectuée et rendue publique tous les ans par le réseau EuroBlight sous forme de cartes interactives: https://agro.au.dk/forskning/internationale-platforme/euroblight/pathogen-monitoring/genotype-map.
Quelques résultats marquants pour la campagne 2024
Une forte pression de maladie
Après une campagne 2022 marquée par une épidémie faible et tardive, et une saison 2023 caractérisée par un développement très atypique du mildiou (épidémie en trois phases), la campagne 2024 a connu une épidémie précoce et forte dans toutes les régions, rappelant la campagne 2021. Les précipitations abondantes et les températures modérées ont en effet favorisé à la fois la croissance des plantes et le développement du parasite, souvent présent dès la levée des plantes. La pression est restée élevée durant toute la campagne, ce qui a imposé une surveillance active et des interventions assez nombreuses en culture.
Des génotypes nouveaux supplantent partout les lignées historiques
La forte épidémie observée en 2024 a favorisé une collecte intensive de cartes d’échantillonage. Ce sont ainsi plus de 300 prélèvements issus de parcelles de production qui ont été analysés, donnant une image assez fiable de la composition des populations sur le territoire national. La plupart de ces cartes proviennent de pomme de terre, seuls quelques échantillons issus de tomate ayant été réceptionnés.
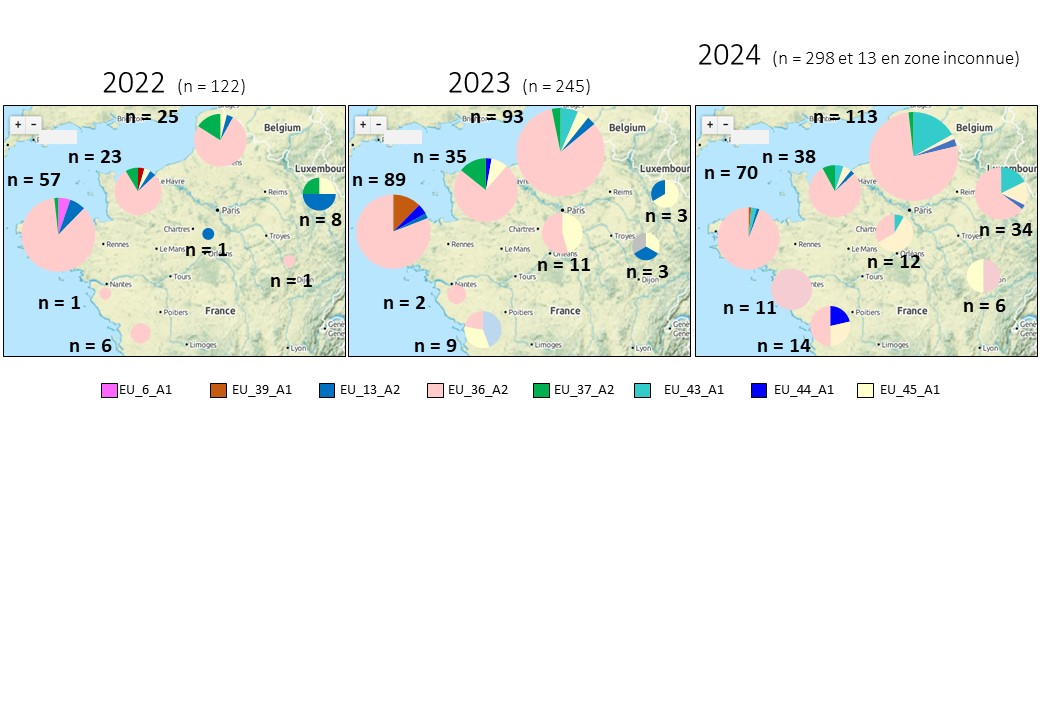
Comme le montre la figure ci-dessous, cinq enseignements principaux peuvent être tirés des données de génotypage obtenues :
- La lignée EU_36_A2 (rose pâle) est désormais très largement majoritaire dans toutes les zones d’échantillonnage ;
- fait marquant de la campagne, la lignée EU_43_A1 progresse, en particulier dans le bassin parisien et en Champagne, mais reste à des fréquences assez faibles (maximum 20 %). Cette lignée a été détectée pour la première fois en Bretagne, mais semble absente des régions au sud de la Loire. Les caractérisations antérieures des souches appartenant à cette lignée établissaient qu’une proportion significative de ces souches étaient résistantes aux fongicides de la famille des CAA (notamment le mandipropamide), et parfois aussi à l’oxathiapiproline. Les analyses sur les échantillons français sont encore en cours, mais nous n’avons reçu aucune mention de perte d’efficacité avérée en France de ces deux matières actives au champ en conditions normales d’utilisation ( alternance systématique de matières actives). La vigilance reste toutefois de mise, dans la mesure où cette lignée est désormais bien établie au Bénélux et au Danemark, où elle cause des difficultés importantes pour la gestion de la lutte ;
- la lignée EU_37_A2 persiste de manière limitée uniquement dans quelques régions (Normandie et dans une moindre mesure Hauts de France), sans doute à relier au recul de l’emploi du fluazinam (matière active à laquelle cette lignée est résistante);
- l’extension à la fois géographique et en fréquence de la lignée EU_45_A1 (jaune pâle) se poursuit : elle est désormais présente dans toutes les régions, à l’exception notable de la Bretagne ;
- la régression, et parfois même la disparition complète des lignées historiques EU_13_A2 (bleu) et EU_6_A1 (rose vif) se poursuit : cette dernière a en effet disparu entièrement des échantillons collectés, alors que la première ne représente désormais que moins de 5 % des échantillons collectés.
Sur tomate, les échantillons de la moitié Nord de la France appartiennent majoritairement aux lignées EU_39_A1 et EU_44_A1, génétiquement assez proches l’une de l’autre. Ces observations confirment une différenciation assez nette entre populations présentes sur pomme de terre d’une part, sur tomate d’autre part, même si des lignées comme EU_36_A2 sont identifiées sur chacun de ces deux hôtes.
|
Fréquence des lignes clonales de Phytophthora infestans dans les différentes régions françaises en 2022, 2023 et 2024. Chaque lignée est représentée par une couleur, et « n » est le nombre d’échantillons analysés pour chaque région. |
Une séparation nette, mais pas absolue, entre populations présentes sur pomme de terre et
sur tomate
Pour la première fois cette année, la campagne d’échantillonnage et d’analyse a été cadencée au cours de la saison de production, en particulier pour répondre à la demande de caractérisation rapide des souches présentes en début d’épidémie. Comme le montre le graphique suivant, les populations initiales sont plus diverses que celles retrouvées dans le courant de l’épidémie, signant l’importance de la sélection exercée par l’hôte et par les pratiques culturales. L’augmentation sensible de la fréquence de EU_43_A1 au cours de la saison correspond ainsi vraisemblablement à l’emploi des CAA pour contrôler les épidémies. Il est donc intéressant de continuer à analyser ces évolutions temporelles en cours de campagne pour ajuster le cas échéant les programmes de lutte .
|
Evolution de la composition des populations de Phytophthora infestans en France au cours de la saison épidémique 2024. Chaque lignée clonale est représentée par une couleur, et « n » est le nombre d’échantillons analysés. |
Quelles conséquences immédiates pour la lutte ?
Caractéristiques des souches françaises. La lignée dominante actuellement, EU_36_A2 ne montre pas de perte de sensibilité aux matières actives autorisées actuellement, à la différence de EU_37_A2 dont la faible sensibilité au fluazinam est désormais bien établie, et de EU_43_A1 dont la plupart des isolats sont reconnus comme résistants aux fongicides de la famille des CAA. Sur le plan du pouvoir pathogène, EU_36_A2 semble un peu plus agressive que EU_13_A2 et EU_37_A2 (les moins agressives), mais moins que la lignée historique EU_6_A1. EU_36_A2 aurait une période de latence (temps écoulé entre l’inoculation et l’apparition des premières spores) assez courte, ce qui peut entrainer un enchainement rapide des cycles épidémiques en conditions climatiques favorables ; ceci pourrait expliquer son expansion rapide lors des quatre dernières saisons. De plus, ces souches semblent assez agressives sur tubercules, ce qui pourrait favoriser leur conservation hivernale (ce que tend à confirmer la fréquence initiale élevée de cette lignée dès le début de campagne). Ces données seront utiles pour accompagner au mieux le déploiement de variétés améliorées pour la résistance au mildiou.
Nous n’avons que très peu d’informations sur les caractéristiques des autres lignées détectées dans le suivi, en particulier la lignée émergente EU_45_A1. Les premiers tests réalisés à INRAE suggèrent toutefois que cette lignée a, comme EU_36_A2,une agressivité intermédiaire entre celles des lignées historiques EU_6_A1 (très agressive) et EU_13_A2 (assez peu agressive). Ces données préliminaires seront complétées par celles obtenues lors des essais mentionnés ci-dessus.
Comparaison avec les populations européennes. Le développement rapide de EU_36_A2, remplaçant EU_13_A2, est visible dans la plupart des pays d’Europe de l’Ouest. Par contre, l’Europe du Nord reste à l’écart de cette tendance, avec le développement d’autres lignées clonales (notamment EU_41_A2 et EU_43_A1) et une présence généralisée de populations sexuées issues d’oospores persistant dans les sols. Ces phénomènes de reproduction sexuée sont apparemment rares en France, ce qui simplifie la lutte car les oospores constituent une source de brassage génétique et de contamination de long terme, difficiles à suivre et à inclure dans les modèles épidémiologiques de prévision des risques.
Il n’est par ailleurs pas anodin de noter que les phénomènes de perte de sensibilité à diverses matières actives tendent à s’accentuer et à s’accélérer : sensibilité faible aux phénylamides chez EU_13_A2, au fluazinam chez EU_37_A2, et résistance au mandipropamide voire à l’oxathiapiproline chez une majorité des souches de EU_43_A1. L’expansion relative de EU_43_A1 est bien moins rapide en France qu’elle ne l’a été au Bénélux ou au Danemark, où cette lignée est devenue rapidement majoritaire. Ceci est sans doute la conséquence d’une stratégie d’emploi plus adéquate en France des matières actives (pas de ‘blocs de traitements’ avec les mêmes familles, alternance stricte, etc…) qui limite visiblement la sélection de ces souches résistantes. Toutefois, cette expansion, même contrôlée, impose la plus grande vigilance quant à la performance de la lutte chimique et à l’élaboration des programmes de traitements.
La surveillance se poursuivra en 2025, avec l’apport de l’application participative VigiMildiou et de nouveaux outils en développement
Les données de surveillance sont un élément de plus en plus important pour le pilotage de la lutte contre le mildiou. Le dispositif déployé ces dernières années sera donc reconduit en 2025 avec la collaboration des mêmes acteurs. Les personnes en régions souhaitant participer à la collecte des échantillons, sur pomme de terre comme sur tomate, peuvent se rapprocher des contacts indiqués ci-dessous, qui leur fourniront des cartes FTA vierges, un mode d’emploi et les instructions pour l’envoi des échantillons. INRAE continuera pour sa part à assurer le génotypage et la synthèse des résultats. Nous tenons à remercier ici l’ensemble des acteurs de terrain, dont l’implication dans la collecte des échantillons pemet de suivre au plus près l’évolution des populations de P. infestans sur l’ensemble du territoire français, mais aussi d’être des intervenants reconnus dans le consortium européen de monitoring des souches de mildiou.
Ce dispositif bénéficie de développements déjà opérationnels ou en cours d’évaluation :
- d’une part, la possibilité de relier très simplement observation du stade épidémique et collecte d’échantillons, grâce à l’application participative VigiMildiou, développée dans le cadre du projet SYNAPTIC (https://ecophytopic.fr/recherche-innovation/proteger/projet-synaptic). Cette application, téléchargeable gratuitement via les plateformes de téléchargement GooglePlay et AppStore (accès direct depuis la page dédiée du portail ephytia : https://ephytia.inra.fr/fr/P/176/VigiMildiou), permet à tous les acteurs, professionnels ou particuliers, de renseigner en quelques secondes et depuis un smartphone la présence de mildiou. Ces observations sont évidemment l’occasion de prélever des échantillons, via les cartes FTA, échantillons qui entreront alors dans le dispositif de surveillance annuelle. C’est donc un moyen pour chacun de contribuer directement à l’effort de suivi des populations de infestans, au bénéfice de tous !
- d’autre part, le développement de méthodes génétiques rapides pour suivre la résistance à plusieurs matières actives (notamment les CAA et l’oxathiapiproline) en routine dans les échantillons collectés. L’application de ces outils aux travaux d’épidémiosurveillance en France devrait intervenir dès 2025, dans le cadre d’un nouveau projet financé via le programme PARSADA ;
- enfin, la mise au point, actuellement en cours de test, d’une méthode d’identification des lignées présentes localement à partir de spores collectées dans l’air via des pièges à spores. La mise au point de cette nouvelle technologie reste à finaliser, mais des premiers essais semblent prometteurs. A suivre donc !
Contacts
Collecte d’échantillons :
- Bretagne - Julie Le Moal (j.lemoal@plantsdebretagne.com); Arnaud Barbary (a.barbary@plantsdebretagne.com)
- Hauts de France, Normandie, Grand Est – Pauline Dewaegeneire (pauline.dewaegeneire@inov3pt.fr) ; Clément Mabire ; Cyril Hannon (c.hannon@arvalis.fr)
- Centre Val de Loire, Pays de la Loire, Nouvelle Aquitaine, Rhônes Alpes – Philippe Laty (philippe.laty@comitecentreetsud.fr); François Ghigonis (f.ghigonis@arvalis.fr)
- Autres régions - Guillaume Saubeau (guillaume.SAUBEAU@florimond-desprez.fr); Denis Gaucher (d.gaucher@arvalis.fr)
Typage, analyse des résultats et rédaction de la note: INRAE, UMR IGEPP, 35653 Le Rheu
- Romain Mabon (romain.mabon@inrae.fr),
- Michèle Guibert (michele.guibert-rolland@inrae.fr),
- Didier Andrivon (didier.andrivon@inrae.fr).





